fitness_over_time <- read_excel("data/Fitness_over_Time.xlsx")Figura 2
Múltiples vías evolutivas para recuperar los costes de fitness
NOTA: Agregar el diagrama con descripcion del analisis.
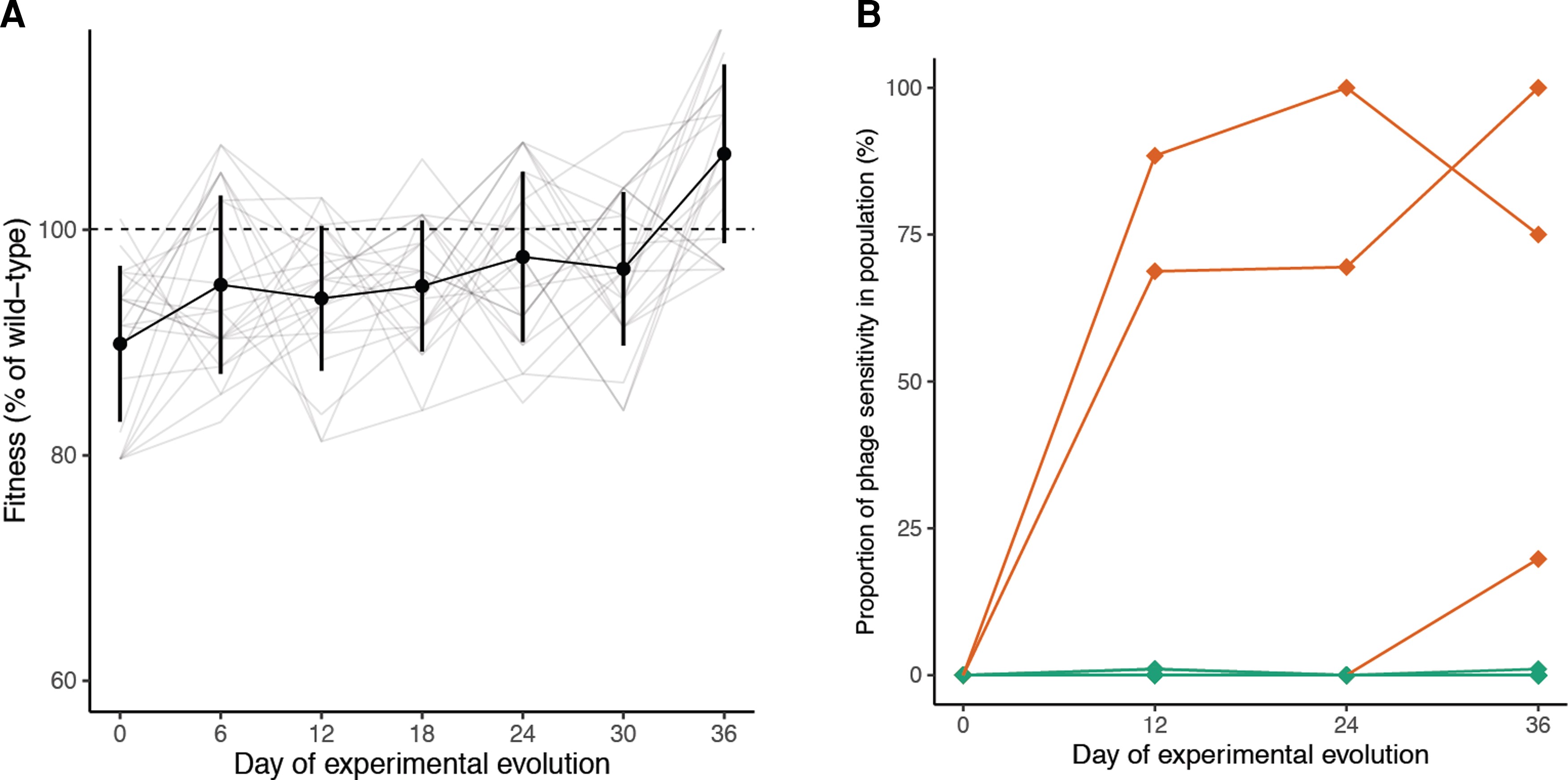
Figure 2A. ¿Cómo cambia la adecuación a lo largo del tiempo?
Cargar los datos
Cargar informacion de “Fitness_over_Time.xlsx” como variable fitness_over_time.
Ajuste de valores
Calcular los valores ajustados de la tasa de crecimiento, controlando por el tipo de bacteria (proveniente de una cepa resistente o no), la posición en la placa y el tiempo en el que se muestreó.
fitness_over_time$fitted <- fitted.values(lm(r~Passage*Type+Column,fitness_over_time))
# Expresar los valores ajustados de la tasa de crecimiento como porcentaje relativo a la tasa de crecimiento del tipo wild-type (iniciada de una cepa sensible a fagos)
for (passage in seq(0,12,2)){
anc_means<-mean(unlist(fitness_over_time[fitness_over_time$Passage==passage & fitness_over_time$Type=="ANC","fitted"]))
fitness_over_time[fitness_over_time$Passage==passage & fitness_over_time$Type=="PR","fitted_percWT"]<-100*fitness_over_time[fitness_over_time$Passage==passage & fitness_over_time$Type=="PR","fitted"]/anc_means
}
# Calcular el promedio y sd del porcentaje de crecimiento de la población de las bacterias iniciadas desde cepas resistents (relativo) por evento de muestreo (passage)
df <- data.frame(aggregate(unlist(fitness_over_time[fitness_over_time$Type=="PR","fitted_percWT"]),
by=list(unlist(fitness_over_time[fitness_over_time$Type=="PR","Passage"])),FUN=mean),
aggregate(unlist(fitness_over_time[fitness_over_time$Type=="PR","fitted_percWT"]),
by=list(unlist(fitness_over_time[fitness_over_time$Type=="PR","Passage"])),FUN=sd)[,2])
colnames(df) <- c("passage","mean","SD") # renombrar columnas
df passage mean SD
1 0 89.89712 6.899891
2 2 95.12769 7.896342
3 4 93.91953 6.402866
4 6 95.01774 5.807250
5 8 97.60042 7.534193
6 10 96.53824 6.803644
7 12 106.73598 7.916310Descripción de la figura
Obtener las tasas de crecimiento de las poblaciones inoculadas con colonias resistentes a los fagos a lo largo de la evolución experimental en ausencia de fagos, calculadas a partir de mediciones de la densidad óptica en un lector de microplacas durante 40 h de crecimiento (n = 22).
Las líneas representan las trayectorias de las poblaciones individuales a lo largo del tiempo, mientras que los puntos indican la aptitud media entre las poblaciones muestreadas el mismo día y las barras de error indican la DE. Los valores se normalizan con respecto a las tasas de crecimiento de las poblaciones de control sensibles a los fagos muestreadas en el mismo punto de la evolución experimental.
Encontraron que con el tiempo la adecuación de las colonias resistentes aumenta hasta ser similar a la del grupo sensible. Esto puede ser porque se pierde la resistencia (y por lo tanto el costo que tiene) o porque se adquieren mutaciones adicionales que compensan ese costo.
Grafica
ggplot() +
geom_point(data=df,aes(x=passage*3,y=mean),size=3) +
geom_errorbar(data=df,aes(x=passage*3,ymin=mean-SD,ymax=mean+SD),width=0,size=1) +
geom_line(data = fitness_over_time, aes(x = Passage*3, y = fitted_percWT, group = Population), linewidth = 0.2, alpha = 0.4)+
geom_line(data=df,aes(x=passage*3,y=mean)) +
theme_classic(base_size=16) +
geom_hline(yintercept=100,linetype="dashed") +
scale_x_continuous(breaks=seq(0,36,6)) +
ylab("Fitness (% of wild-type)") + xlab("Day of experimental evolution") +
coord_cartesian(ylim=c(60,115))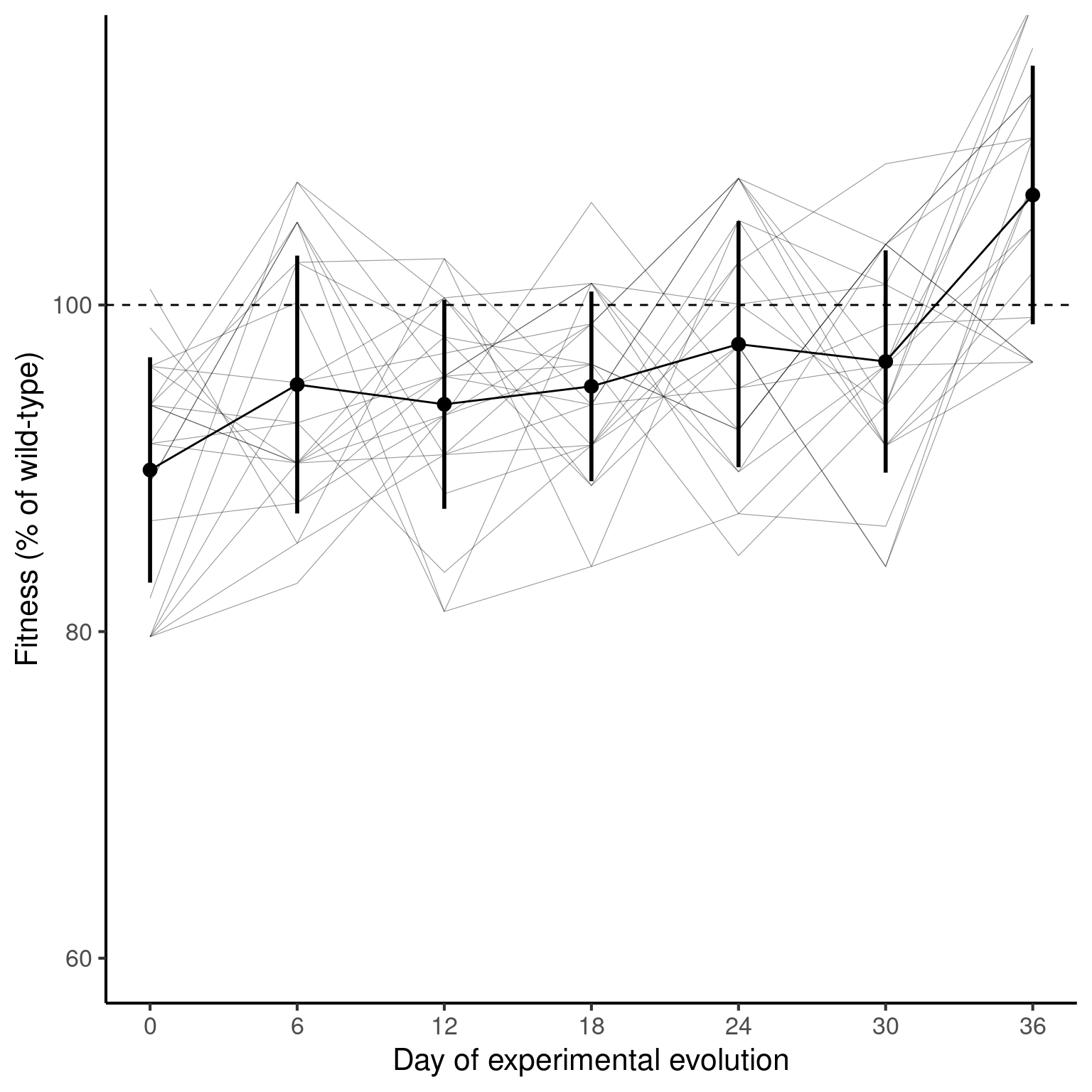
Figure 2B. ¿Cómo cambia la proporción de colonias resistentes en el tiempo?
NOTA: Abri el excel y corre un error de compatibilidad.
Cargar los datos
Cargar informacion de “Resistance_over_Time.xlsx” como variable res_over_time.
res_over_time <- read_excel("data/Resistance_over_Time.xlsx", sheet = "Data")Transformación de los datos en formato largo
res_long <- melt(res_over_time, id.vars = "isolate", measure.vars = c("Prop_S_P0","Prop_S_P4","Prop_S_P8","Prop_S_P12"))
res_long$variable <- as.character(res_long$variable) # Conversión de la columna variable a caracteres:
res_long$passage <- as.numeric(substr(res_long$variable,9,nchar(res_long$variable)))
# Nota: Excluye las poblaciones con colonias que parecían sensibles en las placas pero que no se veían afectadas por el fago en las curvas de crecimiento.
res_long <- res_long[!res_long$isolate %in% c("FMS6","SNK6"),] Descripción de la figura
Proporción de colonias de cada población que se calificaron como sensibles a los fagos a lo largo del tiempo, según lo indicado por la interrupción del crecimiento bacteriano al encontrarse con los fagos en una placa de agar (n = 22 poblaciones con 96 colonias muestreadas por población). Las líneas naranjas indican las poblaciones de las que se obtuvieron colonias sensibles.
Grafica
Al final encuentran que la mayoría de las colonias se mantienen resistentes. Unicamente 5 parecían ser sensibles a los fagos (de esas 2 las excluimos porque el crecimiento no se detuvo cuando fueron expuestas a los fagos). Por lo tanto, solo 3 colonias pierden la resistencia (líneas naranjas).
ggplot(res_long, aes(passage*3,value*100,group=isolate,color=(isolate%in%c("SNK7","QAC5","VCM4")))) +
geom_point(size=4.5,shape=18) +
geom_line(linewidth=0.8)+
theme_classic() +
guides(color=F)+
scale_color_brewer(palette="Dark2") +
scale_x_continuous(breaks=c(0,12,24,36)) +
theme_classic(base_size=16) +
ylab("Proportion of phage sensitivity in population (%)") +
xlab("Day of experimental evolution") +
theme(axis.title.y=element_text(size=14))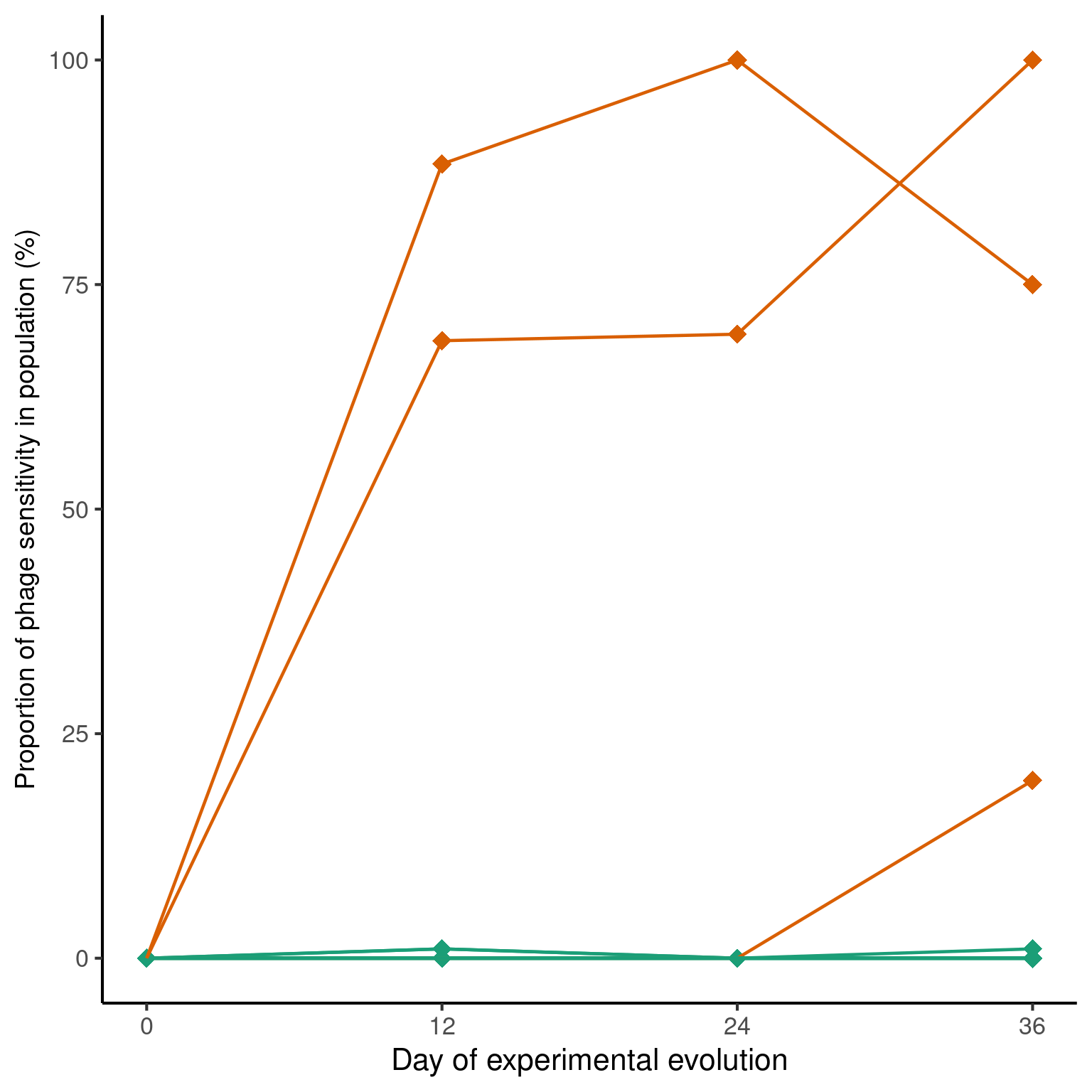
¿Será que el aumento en la adecuación que se observa con el tiempo se debe a la pérdida de la resistencia?
A pesar de que la adecuación si cambió (aumentó) en el tiempo, no hay diferencias significativas entre las poblaciones que se mantuvieron resistentes y las poblaciones que perdieron la resistencia. Por lo tanto, posiblemente las primeras adquirieron nuevas mutaciones que redujeron los costos de adecuación.
fitness_over_time[fitness_over_time$Population %in% c("SNK7","QAC5","VCM4"),"Outcome"] <- "S"
fitness_over_time[!fitness_over_time$Population %in% c("SNK7","QAC5","VCM4"),"Outcome"] <- "R"
anova(lmer(fitted_percWT~Passage*Outcome+(1|Population),fitness_over_time)) Type III Analysis of Variance Table with Satterthwaite's method
Sum Sq Mean Sq NumDF DenDF F value Pr(>F)
Passage 1794.51 1794.51 1 149 32.8548 5.332e-08 ***
Outcome 33.76 33.76 1 149 0.6181 0.433
Passage:Outcome 117.22 117.22 1 149 2.1462 0.145
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1¿Será que las resistencias más costosas tienen más probabilidad de terminar siendo sensibles a los fagos?
Una de sus hipótesis era que las resistencias más costosas (en la figura 1B aquellas que tuvieran tasas de crecimiento más lento) iban a tener mayor probabilidad de perder la resistencia. Por lo tanto comparan el promedio de la tasa de crecimiento original por el resultado al final del experimento pero encuentran que no hay diferencias en la tasa de crecimiento original y la pérdida de resistencia.
load("data/costs_agg.RData")
costs_agg[costs_agg$Population%in%c("SNK7","QAC5","VCM4"),"Outcome"] <- "S"
costs_agg[!costs_agg$Population%in%c("SNK7","QAC5","VCM4"),"Outcome"] <- "R"
t.test(fitted_percWT~Outcome, costs_agg)
Welch Two Sample t-test
data: fitted_percWT by Outcome
t = 1.8179, df = 4.3098, p-value = 0.1381
alternative hypothesis: true difference in means between group R and group S is not equal to 0
95 percent confidence interval:
-2.034388 10.424968
sample estimates:
mean in group R mean in group S
90.49770 86.30241 Observaciones que requieren más investigación
Las poblaciones que adquirieron resistencia por medio de deleciones grandes no perdieron la resistencia
Tres poblaciones que adquirieron resistencia por medio de la misma mutación tuvieron la misma ruta evolutiva (se mantienen resistentes)
Debido a que solo tres colonias perdieron la resistencia no se puede saber si esto estas observaciones tienen alguna relevancia.