library(ggplot2) # Generar gráficos
library(viridis) # Generar paleta de coloresFigura 4
Contingencia histórica en la evolución del genoma
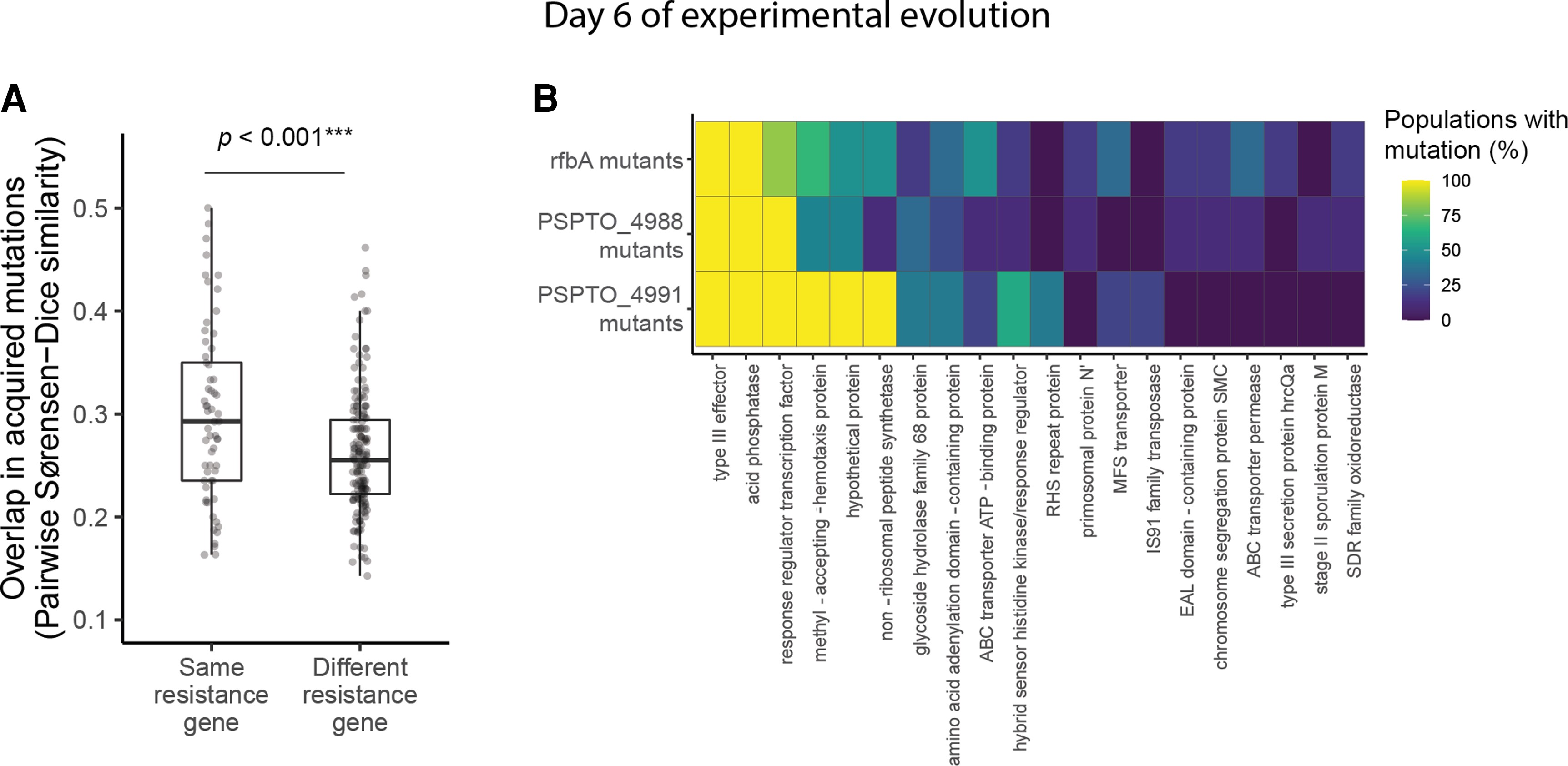
Figura 4 A y 4 B
Para generar las figuras 4A y 4B se tuvieron que seguir varios pasos para procesar los datos, por simplicidad, sólo presentaremos un esquema de los pasos que se siguieron y tomaremos las variables relevantes para generar las figuras.
Esquema
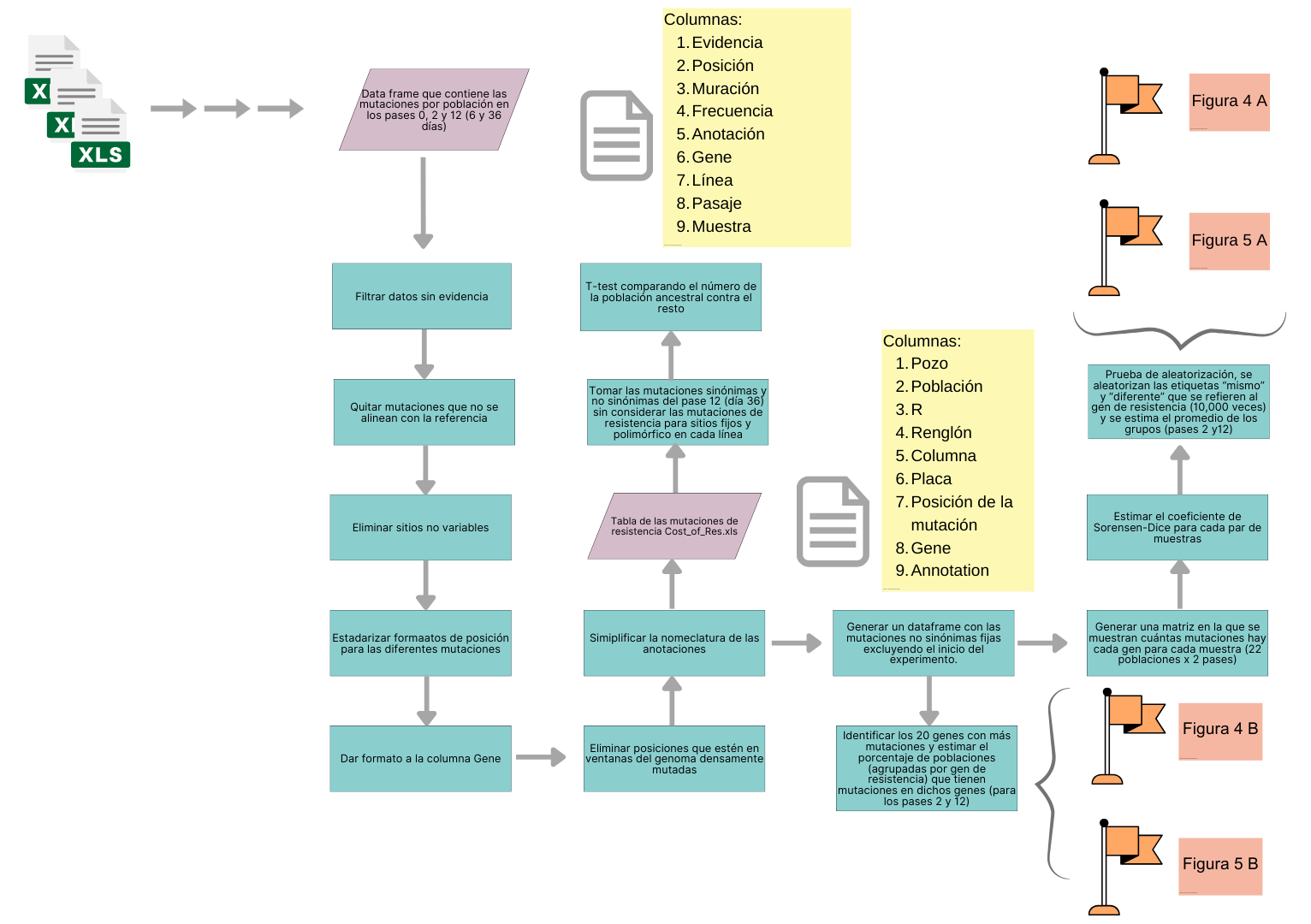
Dificultades
- Los paquetes no estabas especificados.
- La carpeta Breseq indicada, en realidad es la carpeta “Mutation tables” presente en el Github.
- El P value no aparecía en la gráfica de la Figura 4A.
En los heatmap:
- eran cuadrados y los valores del porcentaje iban de 0, 50, 100.
- Los colores no correspondían.
- Los nombres de las mutantes no correspondían.
- El código para las figuras 4 y 5 estaban juntos.
Paquetes
Los paquetes que vamos a cargar son necesarios para las gráficas:
Cargar objetos
Los objetos y variables que cargamos y ocuparemos para esta figura son:
test_gene_df_day6: Data frame con 2 columnas:sim_coefylabel
annotation_day6: Guarda el texto para el p valor.
breseq_gene_props_day6: Data frame con 3 columnas:top_gene,res_geneypercent_pops
load("data/ReproHack_data_figures4_5.RData")Es importante notar que la figura 4 muestra datos para el pase 2, es decir para el día 6 del experimento. Para obetener la figurs 4A se generar boxplots usang ggplot2. Los datos graficados son los de nuestro df: test_gene_df_day6. En este df encontramos los coeficientes de similitud a pares de las 22 poblaciones, la etiquetas nos muestra si tienen la mutación que confiere resistencia en el mismo o en diferente gen. Para colocar el pvalue del test de alotearización se cargó el objeto annotation_day6
p_day6 <- ggplot(test_gene_df_day6, aes(label, sim_coef)) +
geom_boxplot(outlier.shape = NA, width = 0.4, size = 0.8) +
geom_jitter(width = 0.05, size = 2, alpha = 0.2) +
theme_classic(base_size = 18) +
xlab("") +
ylab("Overlap in acquired mutations\n(Pairwise Sørensen-Dice similarity)") +
theme(panel.spacing = unit(3, "lines"),
strip.text = element_text(size = 18, face = "bold")) +
ylim(0.1, 0.55) +
annotate("text", x = 1.5, y = 0.55, label = annotation_day6, size = 6, hjust = 0.5, vjust = 1.5, color = "black")
print(p_day6)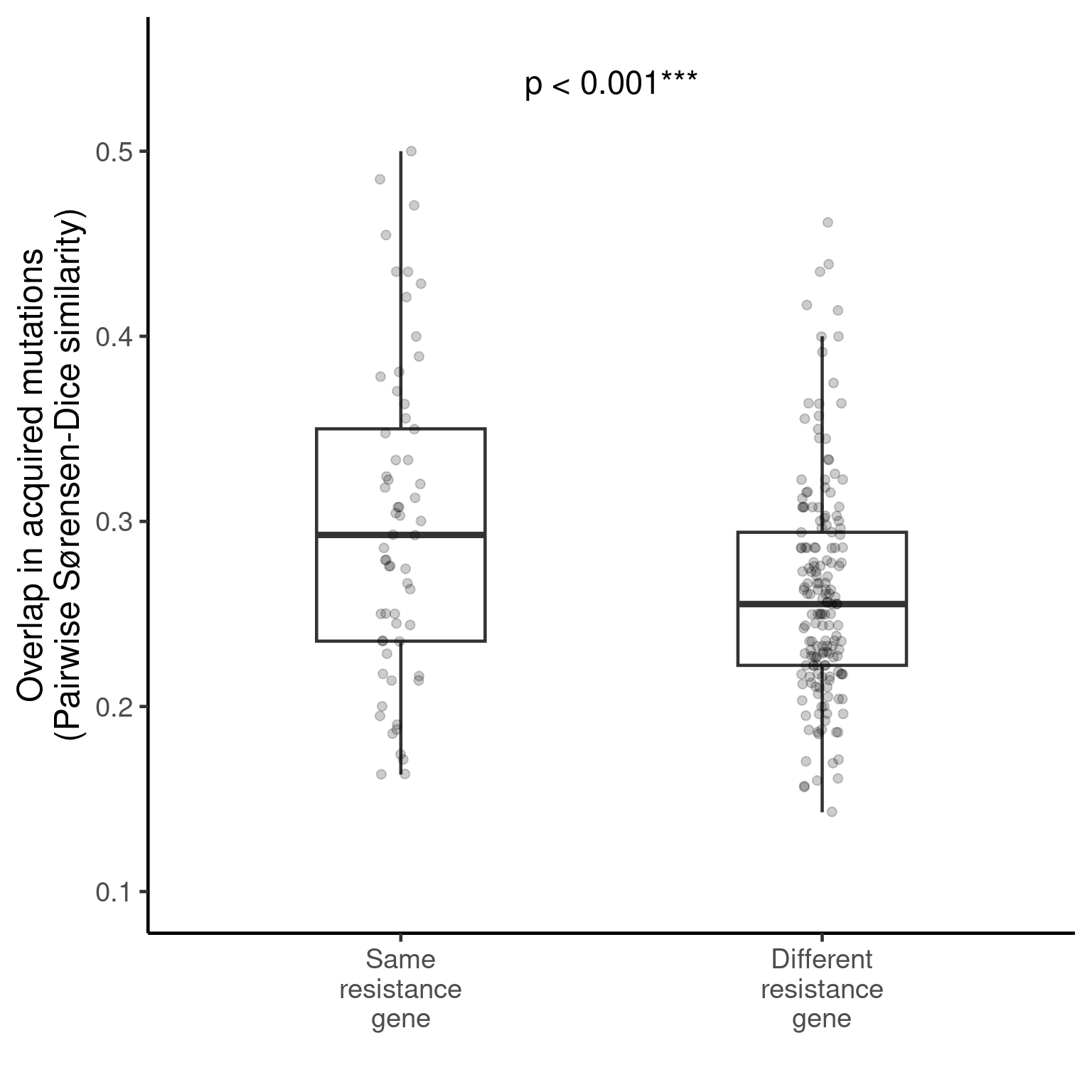
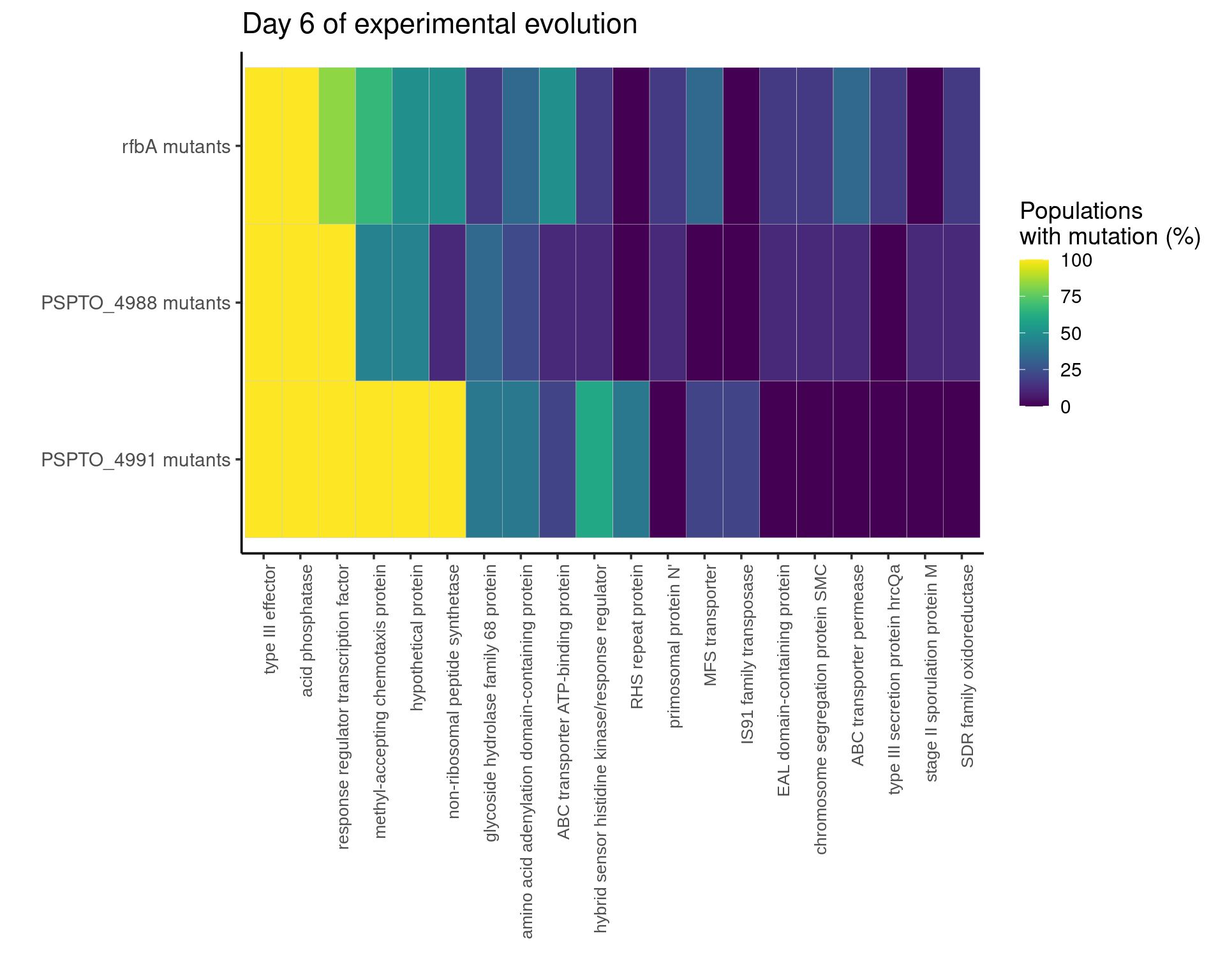
Para el heatmap de las figura 4B usamos ggplot. Nuestra información la tomamos del objeto: breseq_gene_props_day6. Este dataframe nos da información sobre el porcentaje de poblaciones (agrupads por gen de resistencia) que tienen mutación en el top20 de genes.
ggplot(breseq_gene_props_day6, aes(top_gene, res_gene, fill = percent_pops * 100)) +
geom_tile(color = "grey80") +
theme_classic(base_size = 14) +
theme(
axis.text.x = element_text(angle = 90, hjust = 1, size = 10, family = "Arial") # Define all adjustments in one call
) +
scale_fill_viridis(
name = "Populations\nwith mutation (%)",
limits = c(0, 100), # Define scale limits
breaks = c(0, 25, 50, 75, 100) # Define specific breaks to display
) +
ylab("") +
xlab("") +
ggtitle("Day 6 of experimental evolution")