replay <- read_excel("data/Replay.xlsx", sheet = "Data")
replay$Prop_S_P12 <- as.numeric(as.character(replay$Prop_S_P12)) # Convert as numeric / Convertir a numericoFigura 3
Repetición de la evolución experimental
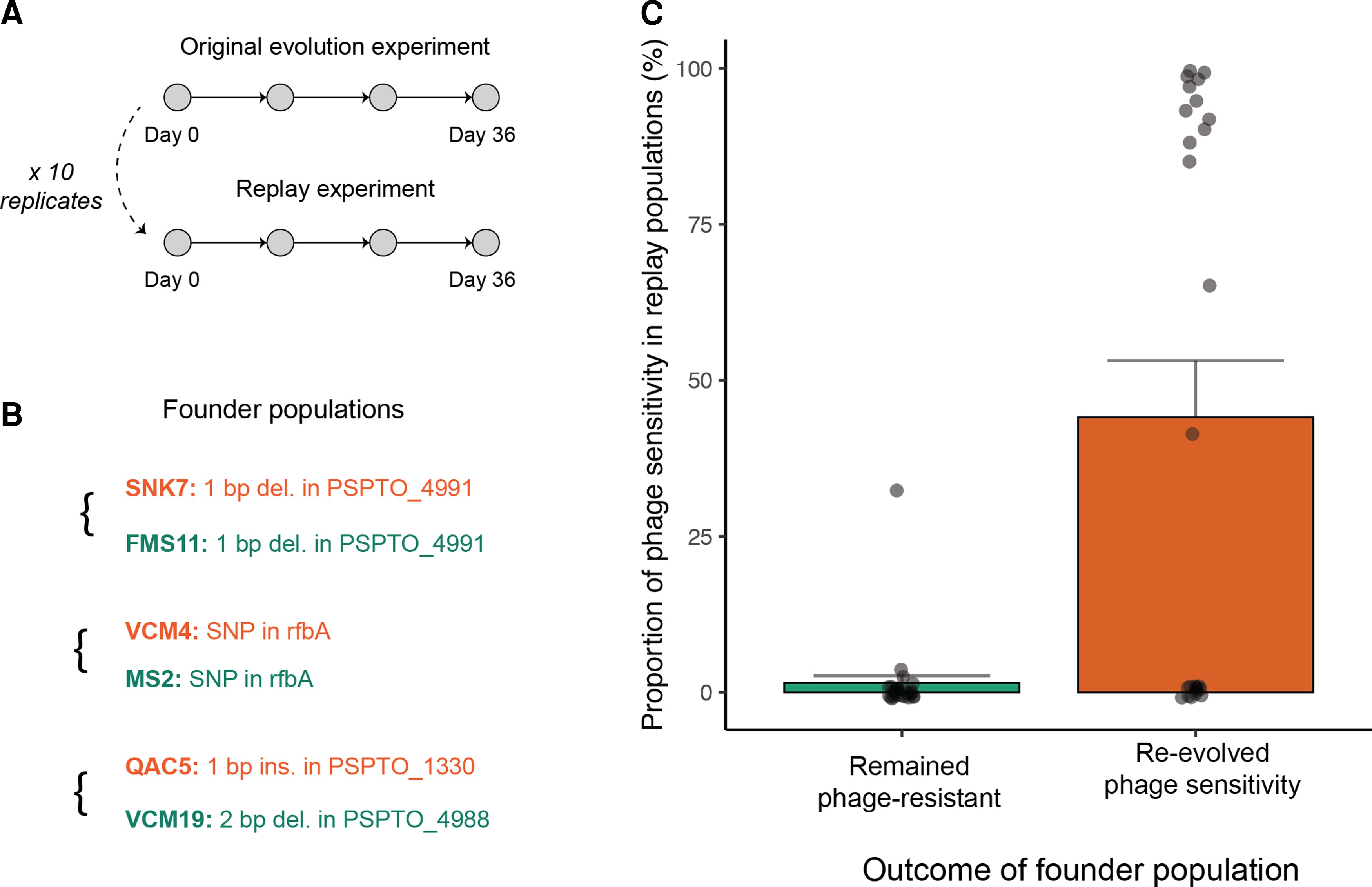
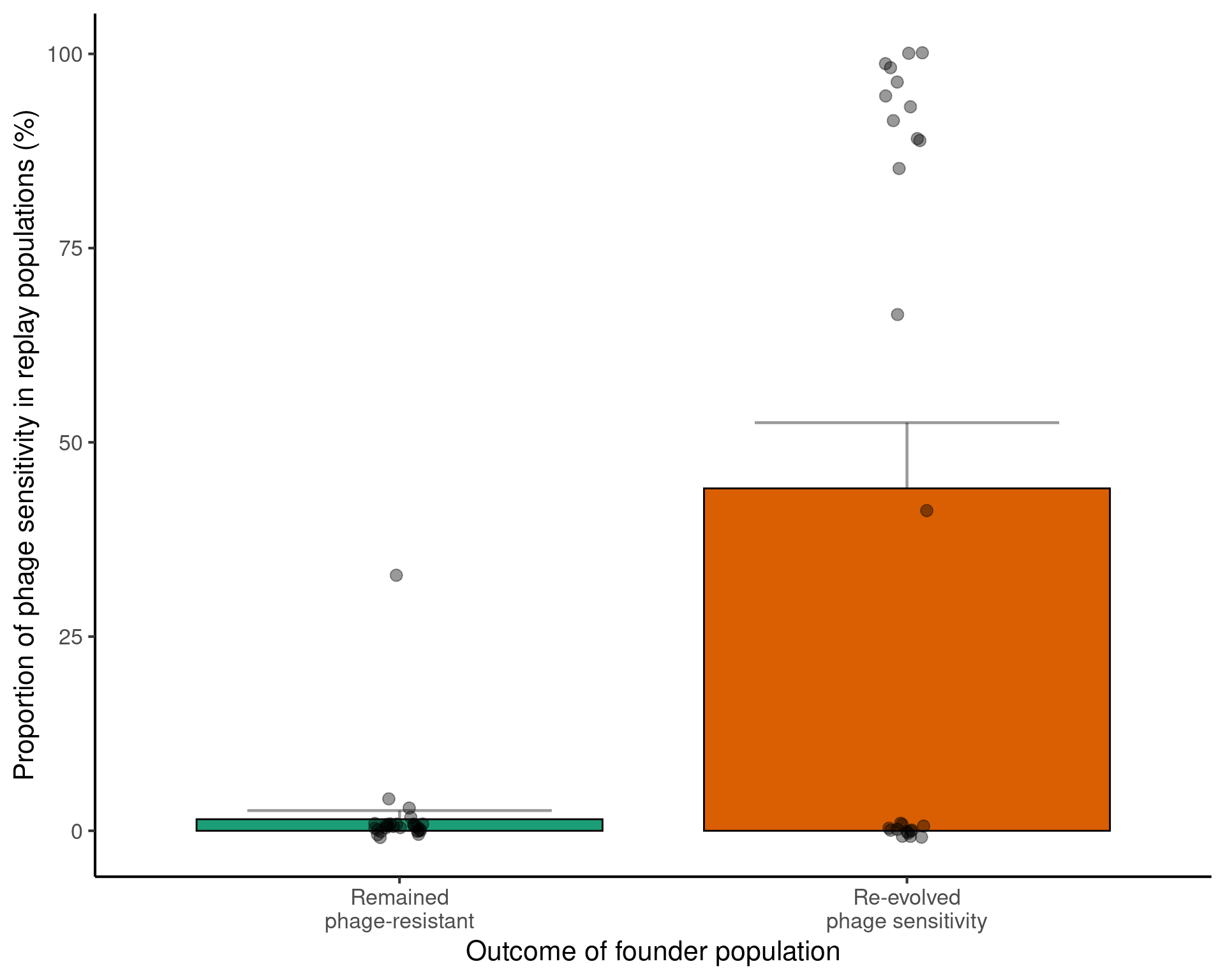
Figura 3C: Los resultados de Resistencia a los fagos en el experimento de repetición reflejan los resultados de su población fundadora
Cargar datos
Cargar informacion de “Replay.xlsx” como variable replay
NOTA: no encuentro la variable
Prop_R_P12, es mas bien la variable se llamabaProp_S_P12.
Calculo de sensibilidad a fagos
Las poblaciones derivadas de un fundador que reevolucionó la sensibilidad tenían más probabilidades de reevolucionar también la sensibilidad
t.test(Prop_S_P12~Founder_outcome, data=replay)
Welch Two Sample t-test
data: Prop_S_P12 by Founder_outcome
t = -4.6491, df = 25.811, p-value = 8.624e-05
alternative hypothesis: true difference in means between group R and group S is not equal to 0
95 percent confidence interval:
-0.6141986 -0.2375002
sample estimates:
mean in group R mean in group S
0.0148947 0.4407441 agg <- data.frame(
aggregate(100*replay$Prop_S_P12, by=list(replay$Founder_outcome), FUN=function(x){mean(x,na.rm=T)}) ,
aggregate(100*replay$Prop_S_P12,by=list(replay$Founder_outcome),FUN=function(x){sd(x,na.rm=T)/sqrt(length(x))}))
# Renombrar columnas
colnames(agg) <- c("Founder_outcome","mean","Founder_2","SE")Descripción de la figura
Proporción de colonias en cada población que se calificaron como sensibles a los fagos después de 36 días en ausencia de fagos, según lo indicado por la interrupción del crecimiento bacteriano al encontrar fagos en una placa de agar (n = 54 poblaciones con 96 colonias muestreadas por población).
Grafica
ggplot() +
geom_errorbar(data=agg,aes(Founder_outcome,ymax=mean+SE,ymin=mean-SE),alpha=0.4,width=0.6,size=0.8) +
stat_summary(data=agg,aes(Founder_outcome,mean,fill=Founder_outcome),geom="bar",fun=mean,width=0.8,color="black") +
geom_jitter(data=replay,aes(Founder_outcome,Prop_S_P12*100),size=3,alpha=0.4,width=0.05,height=1) +
theme_classic(base_size=16) +
scale_fill_brewer(palette="Dark2") +
scale_x_discrete(labels = c("R" = "Remained\nphage-resistant","S"="Re-evolved\nphage sensitivity"))+
guides(fill=F) +
xlab("Outcome of founder population") + ylab("Proportion of phage sensitivity in replay populations (%)")