library(ggplot2) # Generar gráficos
library(viridis) # Generar paleta de coloresFigura 5
El efecto de la historia en la evolución del genoma disminuye con el tiempo
NOTA: Agregar el diagrama con descripcion del analisis.
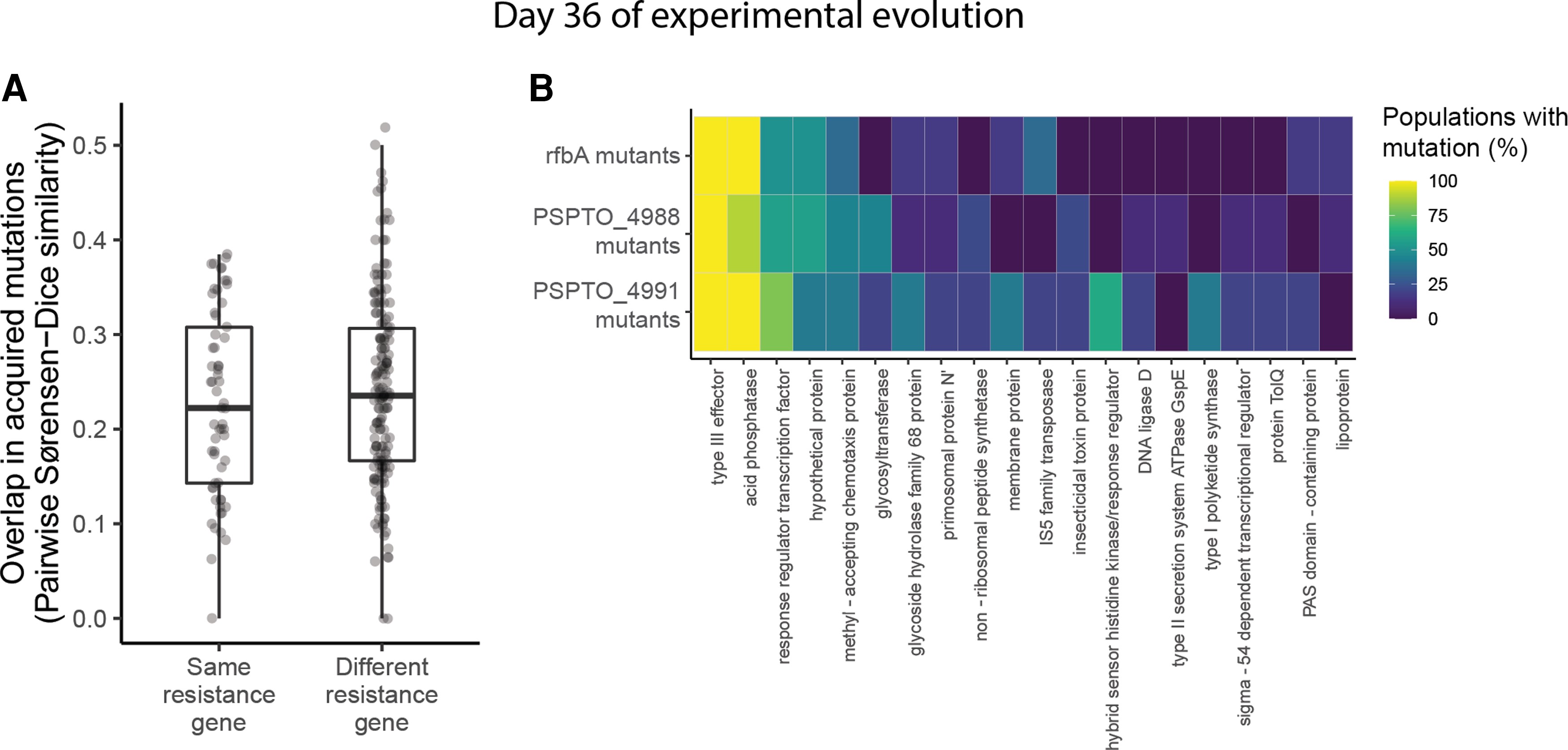
Figura 5 A y 5 B
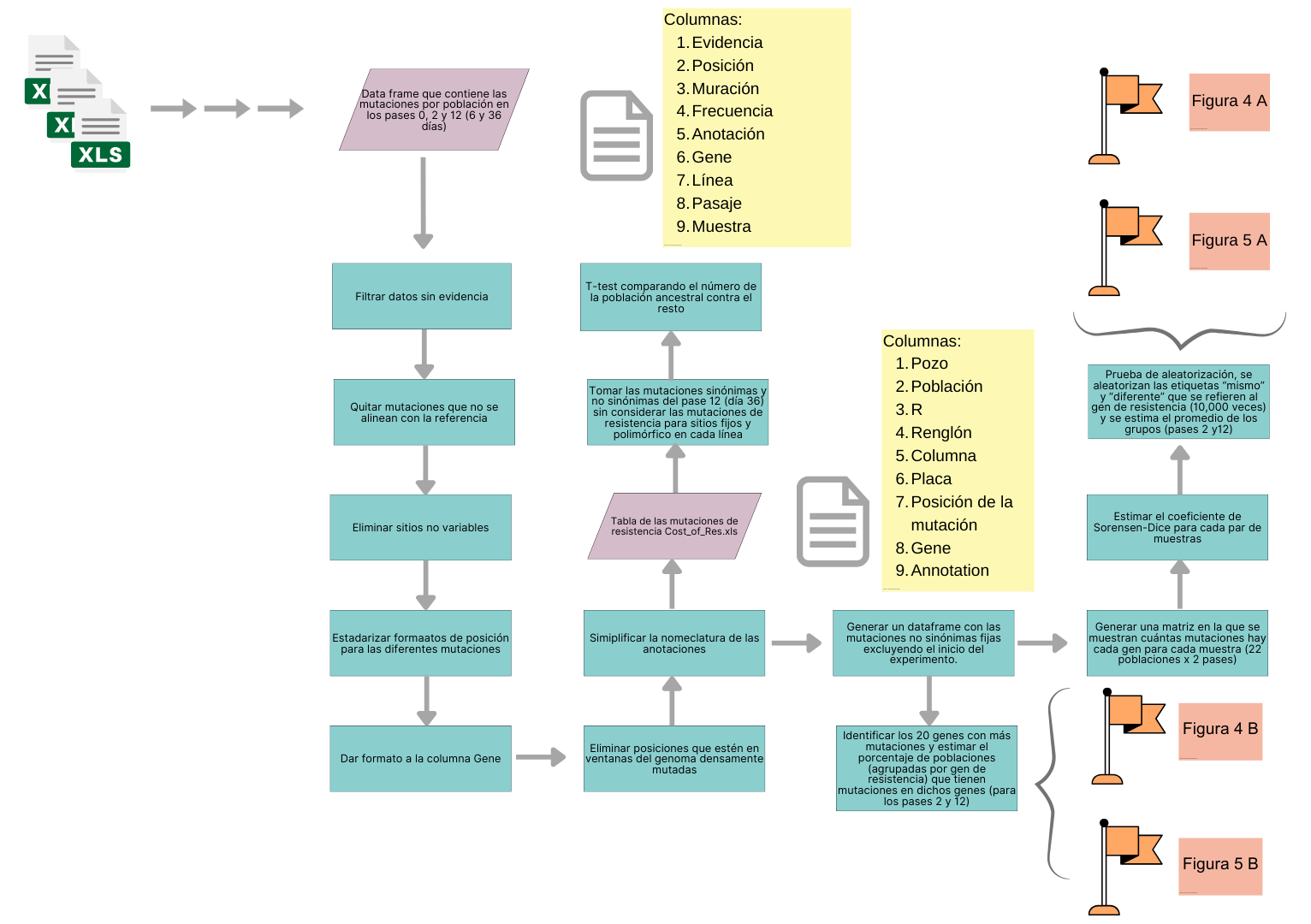
Para generar las figuras 5A y 5B se tuvieron que seguir varios pasos para procesar los datos, por simplicidad, sólo presentaremos un esquema de los pasos que se siguieron y tomaremos las variables relevantes para generar las figuras.
Esquema
Los paquetes que vamos a cargar son necesarios para las gráficas
Paquetes
Cargar objetos
Los objetos y variables que cargamos y ocuparemos para esta figura son: 1. test_gene_df_day36 # Data frame con 2 columnas: sim_coef y label 3. breseq_gene_props_day36 #Data frame con 3 columnas: top_gene, res_gene y percent_pops
load("data/ReproHack_data_figures4_5.RData")Es importante notar que la figura 5 muestra datos para el pase 12, es decir para el día 36 del experimento. Para obetener la figurs 4A se generar boxplots usang ggplot2. Los datos graficados son los de nuestro df: test_gene_df_day36. En este df encontramos los coeficientes de similitud a pares de las 22 poblaciones, la etiquetas nos muestra si tienen la mutación que confiere resistencia en el mismo o en diferente gen.
p_day36 <- ggplot(test_gene_df_day36, aes(label, sim_coef)) +
geom_boxplot(outlier.shape = NA, width = 0.4, size = 0.8) +
geom_jitter(width = 0.05, size = 2, alpha = 0.2) +
theme_classic(base_size = 18) +
xlab("") +
ylab("Overlap in acquired mutations\n(Paired Sørensen-Dice similarity)") +
theme(panel.spacing = unit(3, "lines"),
strip.text = element_text(size = 18, face = "bold")) +
ylim(0.1, 0.55)
print(p_day36)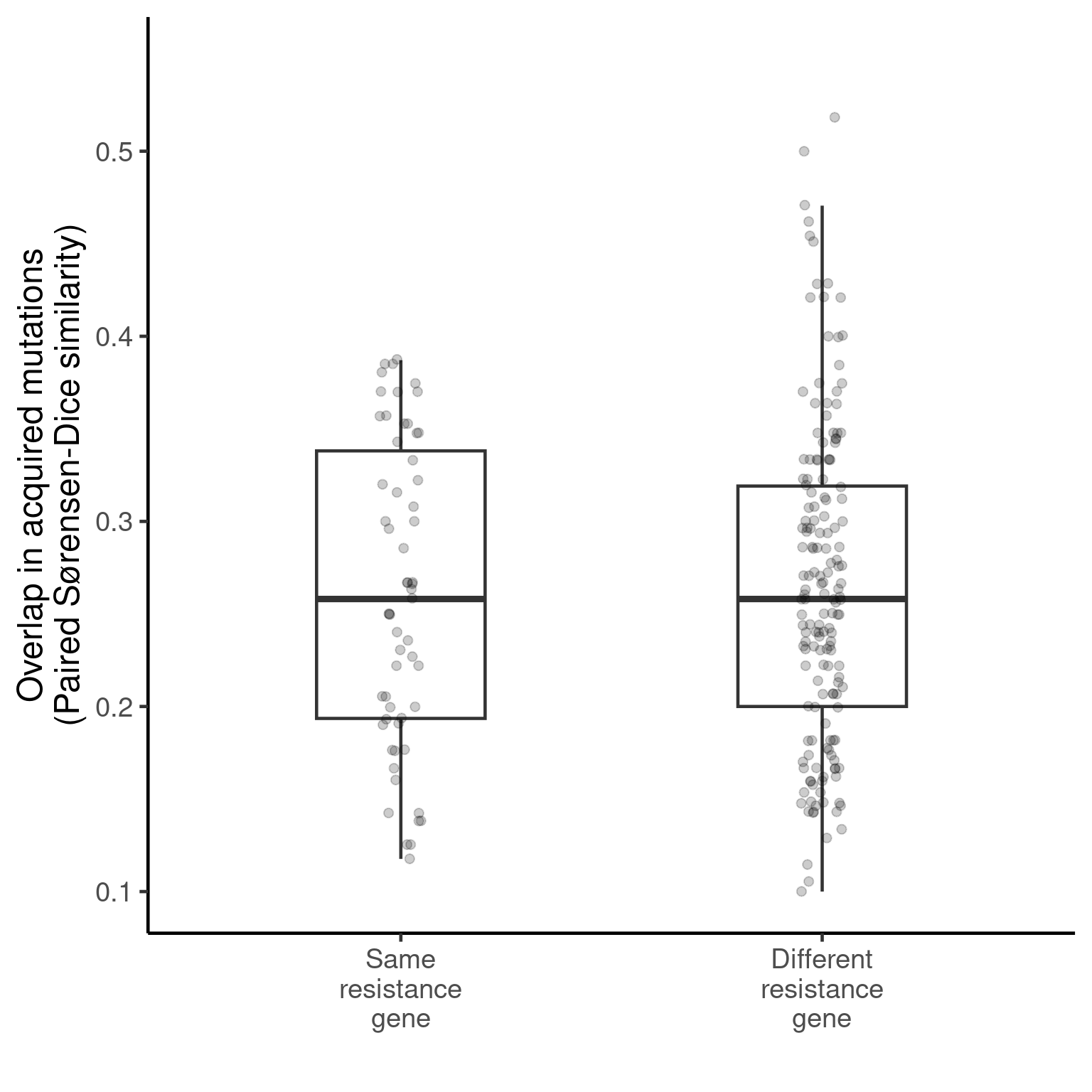
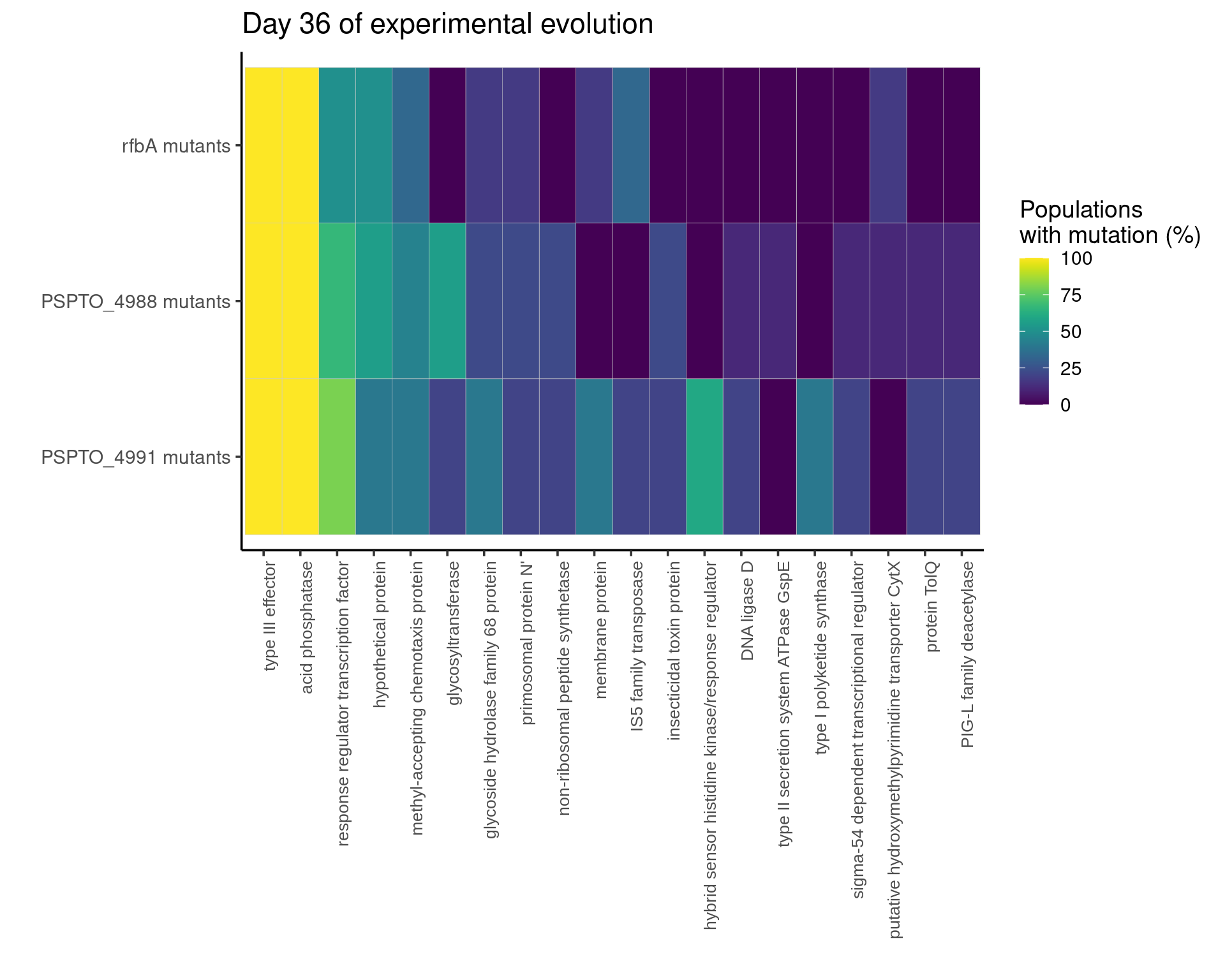
Para el heatmap de las figura 5B usamos ggplot. Nuestra información la tomamos del objeto: breseq_gene_props_day36. Este dataframe nos da información sobre el porcentaje de poblaciones (agrupads por gen de resistencia) que tienen mutación en el top20 de genes.
ggplot(breseq_gene_props_day36, aes(top_gene, res_gene, fill = percent_pops * 100)) +
geom_tile(color = "grey80") +
theme_classic(base_size = 14) +
theme(axis.text.x = element_text(angle = 90, hjust = 1, size = 10, family = "Arial")) +
scale_fill_viridis(name = "Populations\nwith mutation (%)", limits = c(0, 100), breaks = c(0, 25, 50, 75, 100)) +
ylab("") +
xlab("") +
ggtitle("Day 36 of experimental evolution")